La Coévolution Rapide Entre Les Bactéries Et Les Bactériophages Favorise L’émergence De Réseaux À Plusieurs Échelles
RESUMÉ
Les bactéries et les bactériophages sont ubiquitaires. Parasites obligatoires des bactéries, les bactériophages modifient fortement la diversité et la dynamique des populations bactériennes. Comprendre l’interaction bactérie-bactériophage, pour prévoir et contrôler leur évolution a de nombreux intérêts en écologie, industrie, et en santé publique. La coexistence des bactéries et des bactériophages repose en partie sur la coévolution antagoniste qui s’établit entre eux.
Nous avons choisi d’étudier les contraintes génétiques limitant la coévolution entre Escherichia coli et du phage PhiX174.
En étudiant la niche écologique du phage PhiX174, nous avons établi que les souches sensibles à ce bactériophage étaient à la fois rares et remarquables par leur diversité phylogénétique. De plus, alors que le lipopolysaccharide (LPS) est le récepteur reconnu par PhiX174, nous avons démontré que le phage PhiX174 peut lyser des bactéries arborant des LPSs très variés, ce qui contraint la population bactérienne à résister autrement qu’en modifiant son récepteur. Puis, en modifiant le génotype de l’hôte bactérien ensemencé avec le phage PhiX174, nous avons profondément altéré la pérennité de la coexistence bactérie-bactériophage. Enfin, nous avons mis en place un protocole de coévolution entre E. coli C et le phage PhiX174 qui nous a permis de discuter de l’évolution de la pression de sélection induite par les bactériophages sur la population bactérienne et de l’existence d’un compromis entre virulence et infectivité.
Il semblerait, en définitive, que les fortes relations épistatiques entre les gènes participant à l’interaction bactérie-bactériophage, sont autant de contraintes génétiques qui s’ajoutent aux contraintes écologiques déjà décrites et limitent la coévolution antagoniste qui s’établit entre E. coli et phage PhiX174.
INTRODUCTION
Dans l’étude bibliographique ci-après, vous allez explorer le monde invisible des microorganismes. Nous retracerons ensemble l’histoire de la découverte des bactéries et des virus. Puis, nous considérerons l’importance écologique de l’interaction bactérie – bactériophage. Nous étudierons, ensuite, la notion de coévolution antagoniste, les modèles théoriques et expérimentaux existants, et les paramètres déterminant l’évolution des deux protagonistes. Enfin, le système expérimental Escherichia coli – phage PhiX174.
Dans un deuxième temps, vous trouverez les résultats obtenus. A savoir l’étude de la niche écologique du phage PhiX174 ; de l’influence du génotype hôte sur la durée de coexistence E. coli – phage PhiX174 ; et du taux d’évolution du phage PhiX174 en coévolution continue avec E. coli.
Enfin, nous discuterons d’une part des contraintes génétiques limitant la coévolution antagoniste entre la bactérie hôte E. coli et le bactériophage parasite PhiX174, et d’autre part des intérêts en écologie, pour l’industrie et la santé publique de l’étude de la coévolution bactérie-bactériophage et de ce qui peut être fait avec le modèle expérimental E. coli – phage PhiX174.
Pour déterminer la niche écologique du bactériophage PhiX174, 783 isolats naturels d’Escherichia coli ont été examinés pour leur susceptibilité.
Les souches sensibles sont diverses en ce qui concerne leur phylogénie et leur lipopolysaccharide (LPS) central, mais elles présentent toutes des phénotypes rugueux. L’analyse des mutants d’E. coli K-12 LPS a révélé que la susceptibilité à PhiX174 résulte d’une convergence phénotypique avec divers défauts moléculaires.
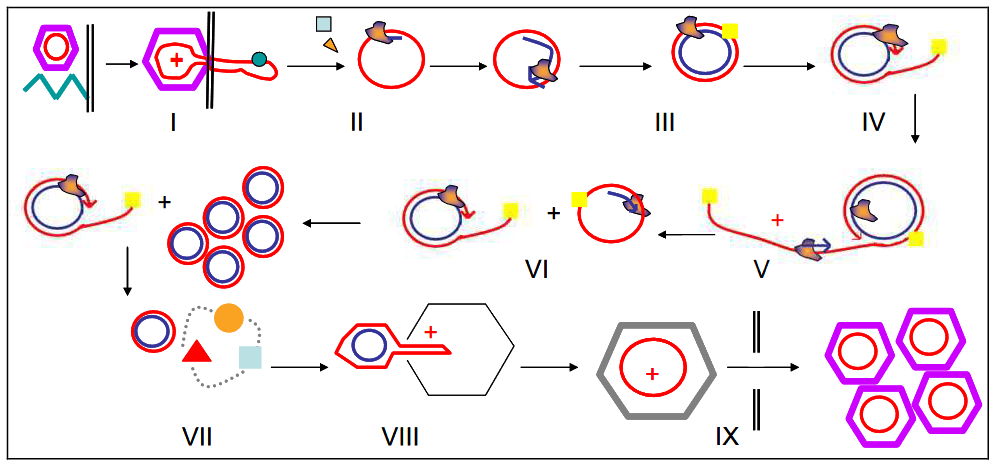
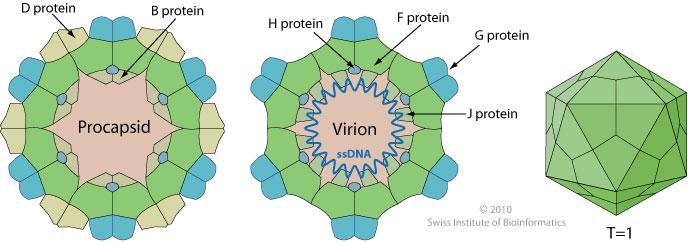
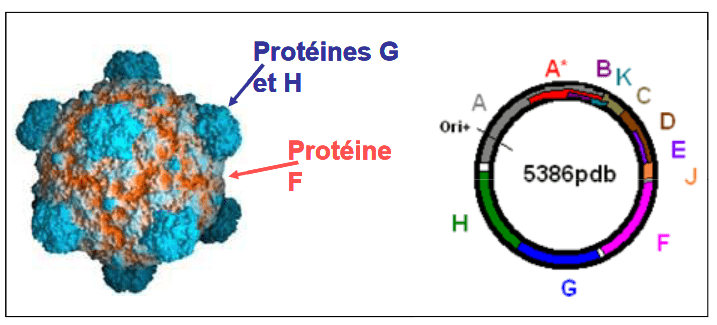
PhiX174 appartient à la famille des bactériophages Microviridae. Il s’agit d’un petit virus icosaédrique non enveloppé ayant un ADN circulaire à brin unique. Depuis son isolement en 1935, PhiX174 a été utilisé dans de nombreuses expériences phares en raison de sa petite taille de génome (5386 nucléotides) et de son statut non pathogène. De plus, comme PhiX174 est un coliphage, il peut être utilisé comme indicateur de contaminations virales ou fécales dans les environnements aquatiques (Organisation internationale de normalisation – ISO 10705-2).
En 1974, Suzuki et al. ont constaté que, bien que l’infection par le phage soit limitée aux bactéries ayant des récepteurs spécifiques, la réplication de l’ADN de PhiX174 peut être soutenue par différentes souches d’Escherichia coli et des bactéries éloignées telles que Pseudomonas aeruginosa. Cela implique que l’étape limitante pour l’infection par PhiX174 est l’entrée et non la réplication ni la lyse. L’hôte de PhiX174 couramment utilisé est la souche dérivée en laboratoire E. coli C, qui a un lipopolysaccharide (LPS) rugueux spécifique reconnu comme le récepteur.
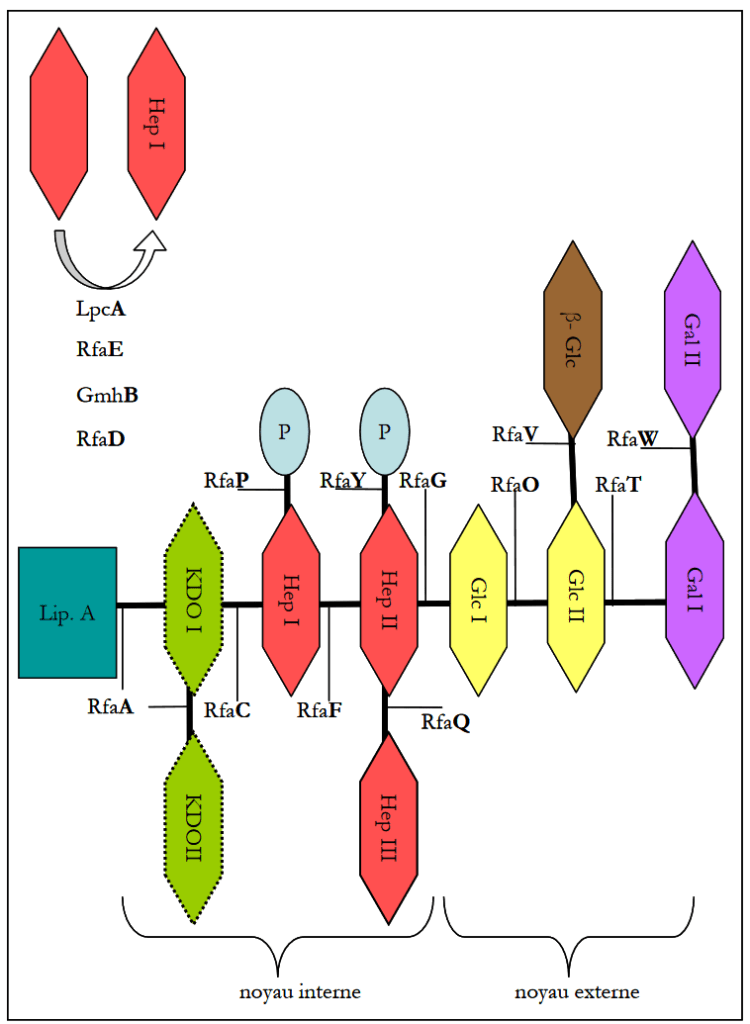
Le LPS est un composant majeur de la membrane externe des entérobactéries, essentiel pour leur stabilité et leur interaction avec le monde extérieur. Il est composé d’un lipide A ancré dans la membrane, d’un noyau oligosaccharidique et peut avoir un polysaccharide (O-antigène) lié à ce noyau. La partie interne du LPS central est hautement conservée chez les bactéries à Gram négatif, tandis que la structure biochimique externe du LPS central est plus diverse. Dans l’espèce E. coli, cinq types externes de noyau ont été décrits : R1, R2, R3, R4 et K-12. E. coli C présente un noyau de type R1. L’étude de sa séquence récemment publiée révèle que son LPS central est pleinement fonctionnel, mais que l’O-antigène est affecté par une insertion IS dans le locus rfb qui génère son phénotype rugueux (numéro d’accession GenBank : CP000946). De manière intéressante, une insertion IS similaire est trouvée dans K-12, le rendant également rugueux mais néanmoins non sensible à PhiX174, ce qui suggère que l’exposition du noyau R1 pourrait être cruciale pour l’infection par PhiX174.
Parmi la collection ECOR, qui est représentative de la diversité génétique de l’ensemble de l’espèce E. coli, jusqu’à 70 % sont de type R1. Cependant, seulement 3 % des souches d’E. coli isolées dans les eaux usées, les selles, l’eau potable ou en laboratoire se sont révélées sensibles à PhiX174.
Pour mieux définir les déterminants moléculaires qui affectent la niche écologique du virus modèle PhiX174, nous avons examiné une vaste collection d’isolats naturels d’E. coli pour la susceptibilité à PhiX174. Nous avons caractérisé les souches sensibles identifiées en fonction de leur groupe phylogénétique, de leur sérotype et de leur type central de lipopolysaccharide (LPS). Nous avons étudié la susceptibilité à PhiX174 des mutants de LPS d’E. coli K-12. Notre analyse a révélé que la sensibilité à PhiX174 est une convergence phénotypique avec des bases moléculaires diverses.
Un créneau restreint pour PhiX174 parmi les isolats naturels d’E. coli
Une collection de 783 isolats naturels d’E. coli a été examinée pour la susceptibilité à PhiX174 : 72 souches ECOR, englobant des souches commensales et pathogènes, provenant d’hôtes humains et animaux, et de différents pays ; 372 souches commensales humaines, de volontaires en bonne santé en France, en Colombie (28), et des souches du Bénin, de Croatie et du Mali isolées à partir de selles d’animaux sauvages ou domestiques français. Des gouttes de cultures d’isolats naturels en culture nocturne ont été déposées sur une gélose supérieure chargée de 107 PFU/mL de PhiX174 (DSMZ4497) et incubées à 33 °C comme dans la sensibilité à PhiX174 des bactéries a été déterminée lorsque celles-ci ne se développaient pas sur une surface ronde et opaque. Nous avons vérifié la sensibilité de ces bactéries qui ne se développaient pas, ou qui formaient une surface ronde irrégulière, avec des gouttes de la solution stock de PhiX174 sur une couche de la culture bactérienne.
Seulement six souches d’E. coli, soit 0,8 %, ont été jugées sensibles à PhiX174. Ce résultat est conforme à des études antérieures. Ces souches proviennent du monde entier (France, Mali et Croatie). Cependant, il semble qu’elles soient toutes commensales d’origine humaine (test exact de Fisher p=0,04). Cette observation pourrait être intéressante car les souches d’E. coli humaines ont peu de spécificités et cela pourrait mettre en évidence une certaine pression de sélection spécifique à l’humain.
Pour approfondir l’étude des exigences de sensibilité à PhiX174, nous avons ensuite cherché des traits génétiques ou phénotypiques qui pourraient expliquer leur sensibilité commune.
Diversité des isolats naturels d’E. coli sensibles à PhiX174
Les types O des souches sensibles ont été obtenus par agglutination avec des antisérums O (Statens Serum Institute, Copenhague, Danemark) et toutes semblaient s’auto-agglutiner (Tableau 1), confirmant que les souches sensibles à PhiX174 sont rugueuses, sans O-antigène lié à leur LPS central. Le groupe phylogénétique et les sous-groupes ont été attribués aux souches sensibles d’E. coli en utilisant la méthode de phylotypage par PCR triplex. Elles appartiennent aux groupes/sous-groupes A1, A0, B1 et B23. La souche de référence d’E. coli C sensible à PhiX174 (DSM13127) appartient au sous-groupe A0. Cela implique que les souches sensibles ne sont pas monophylétiques et que le phénotype O-rugueux sensible à PhiX174 est apparu indépendamment à plusieurs reprises. Cela soutient davantage notre hypothèse selon laquelle l’entrée est l’étape limitante dans la propagation de PhiX174, et cela implique que la sensibilité à PhiX174 pourrait survenir par convergence moléculaire. Étant donné que les souches O-rugueuses exposent le noyau de leur LPS, nous avons vérifié si les souches sensibles partagent un LPS central commun.
Diversité de la structure centrale du LPS des isolats naturels d’E. coli sensibles à PhiX174
De nombreux enzymes participent à la synthèse du LPS. Les gènes codant pour la biosynthèse du noyau du LPS se trouvent principalement dans le locus waa (ou rfa), organisés en trois opérons gmhD, waaQ et waaA, avec trois autres gènes rfaE, lpcA, gmbB répartis plus loin sur le génome. Cinq types de noyau externe du LPS d’E. coli ont été décrits (R1, R2, R3, R4 et K-12) ; ils correspondent à différentes compositions et séquences de l’opéron waaQ. Une méthode PCR, avec des amorces du locus rfa, a été utilisée pour différencier les cinq types de noyau du LPS. Les isolats naturels d’E. coli sensibles à PhiX174 ont été trouvés pour avoir un noyau R1, R2, voire même R3. Pour la souche VDG401, les PCR pour R1 et R2 étaient toutes deux positives. Cela pourrait correspondre à un type de noyau du LPS non décrit.
Une fois de plus, ces souches sensibles se distinguent par leur diversité. Étant donné qu’il semble qu’elles aient différents opérons rfa, différentes mutations pourraient expliquer leur phénotype rugueux commun, et donc leur sensibilité à PhiX174. Pour le confirmer, nous avons étudié l’impact des knockouts de différents gènes impliqués dans la synthèse du noyau du LPS de la bactérie modèle E. coli K-12 sur sa susceptibilité à PhiX174.
Sensibilité à PhiX174 des mutants du noyau LPS d’E. coli K-12
E. coli K-12 présente un type de noyau LPS K-12, a un phénotype rugueux, et est résistant à PhiX174. La sensibilité à PhiX174 des mutants à knockout génique unique rfaB, C, D, E, F, G, H, I, J, L, P, Q, S, T, Y, Z, gmhB et lpcA a été testée en utilisant la collection KEIO. Ils étaient tous résistants à la lyse par PhiX174, à l’exception de rfaB, rfaC, rfaD, rfaE, gmhB et lpcA.
Selon la littérature, les suppressions de rfaC, rfaE, lpcA confèrent un phénotype “deep rough” aux bactéries, empêchant la fixation du premier heptose du noyau interne du LPS. De même, l’enzyme GmhB participe à la biosynthèse du premier heptose. Néanmoins, son activité peut être partiellement complémentée par une autre enzyme. La suppression de rfaD génère un LPS hétérogène, sans heptose ou avec une forme incorrecte de l’heptose. La mutation rfaB modifie le noyau externe. Étant donné que le noyau interne est hautement conservé dans l’espèce E. coli, contrairement au noyau externe, nous nous sommes concentrés sur les mutations “deep rough”. Il est intéressant de noter que grâce à ces mutants sensibles d’E. coli K-12, les interactions bactérie/bactériophage entre E. coli et PhiX174 peuvent désormais être étudiées en profondeur, compte tenu de l’énorme quantité de connaissances accumulées sur E. coli K-12.
Flexibilité du LPS des souches sensibles à PhiX174
La complémentation des suppressions géniques rfaC, rfaE, gmhB et lpcA d’E. coli K-12 avec leurs plasmides ASKA respectifs, induits par le thio-galactoside d’isopropyle-beta (concentration finale de 0,1 μM), a rétabli la résistance. Cependant, lorsqu’ils étaient électroporés et induits dans les isolats naturels d’E. coli sensibles à PhiX174, aucun n’est devenu résistant à PhiX174. Cela implique que leur sensibilité ne peut pas être expliquée par une mutation délétère unique dans l’un de ces gènes. De plus, cela suggère que des mutations dans les parties plus diverses du LPS central pourraient être également responsables de la sensibilité à PhiX174. Ainsi, la sensibilité à PhiX174 peut être soutenue par une grande variété de différents défauts moléculaires affectant le LPS, que ce soit chez E. coli K-12 ou chez les isolats naturels d’E. coli.
Cette flexibilité dans la reconnaissance par PhiX174 a déjà été décrite. Différentes mutations sur des souches sensibles O-rugueuses de type R1 (dérivées d’E. coli C) ont eu différents impacts sur leur reconnaissance par PhiX174. Par exemple, la suppression du galactose terminal rend les bactéries résistantes, tandis que la suppression du glucose terminal (rfaV) ne le fait pas, mais cette dernière suppression diminue leur sensibilité. De même, la suppression du troisième heptose du noyau interne du LPS O-rugueux de type R1 (rfaQ) n’affecte pas la sensibilité à PhiX174. Ainsi, le noyau externe du LPS O-rugueux de type R1 peut tolérer quelques mutations sans rendre les bactéries résistantes. De plus, Salmonella typhimurium peut être sensible à PhiX174 malgré son noyau LPS non-R1, qui porte des hexoses et des ramifications différents. À partir de toutes ces observations, il a été suggéré que le processus de reconnaissance et d’adsorption de PhiX174 dépend de la conformation globale du LPS plutôt que de sa composition précise. La reconnaissance des hôtes potentiels par PhiX174 présente donc une certaine flexibilité. Ici, nous montrons que cette flexibilité est encore plus grande, car nous montrons que les souches de type de noyau R2 ou R3, ainsi que les mutants “deep-rough” d’E. coli K-12, peuvent également être sensibles à PhiX174, et donc le noyau LPS peut être complet, modifié ou même totalement tronqué.
Implications évolutives
En considérant que certaines souches “deep-rough” d’E. coli K-12 sont sensibles à PhiX174, nous suggérons ici que ces souches “deep-rough” sont fortement désavantagées. En effet, elles se caractérisent in vitro par un taux de croissance réduit, une hypersensibilité aux détergents et aux antibiotiques hydrophobes, et sont sensibles à certains phages spécifiques rugueux . De plus, elles sont affectées dans leur mouvement, dans leur capacité à conjuguer, et elles présentent un phénotype de virulence atténué. Tous ces inconvénients rendent les souches “deep-rough” contre-sélectionnées à long terme et donc rares. La variabilité génétique que nous avons observée dans les isolats naturels sensibles confirme davantage l’idée que la sensibilité à PhiX174 émerge de novo par un défaut moléculaire et est contre-sélectionnée à long terme. En effet, il n’y a pas de groupe monophylétique substantiel d’isolats naturels d’E. coli O-rugueux qui soutiendrait une aptitude équitable à long terme pour un tel phénotype. La probable faible aptitude à long terme de ces souches suggère que la niche de PhiX174 est composée de souches ayant récemment perdu leur intégrité du LPS, avec des arrière-plans génétiques variables et des structures de LPS variables. Cela oblige PhiX174 à faire face à une très grande diversité de types d’hôtes dans la nature. Cela pourrait expliquer en partie pourquoi la plupart des adaptations du phage observées sur des souches de laboratoire étaient associées à l’adsorption, car le PhiX174 sauvage doit probablement être généraliste.

Taux d’évolution virale dans un système coévolutif Escherichia coli C – PhiX174
Les paramètres environnementaux abiotiques et biotiques influent sur les forces sélectives agissant sur les populations microbiennes. Dans cette étude, nous avons examiné l’impact des interactions de coévolution antagoniste entre Escherichia coli C et PhiX174 sur le taux d’évolution moléculaire virale. Nous avons considéré que le taux d’évolution virale refléterait la dynamique de la coévolution et donc l’intensité de la pression sélective correspondante sur les bactéries. Nous avons constaté que la plupart des adaptations se produisaient dans la partie du génome du phage impliquée dans l’interaction hôte-parasite. Cependant, nous n’avons pas pu détecter de différences claires dans les taux d’évolution entre les virus en coévolution continue et ceux faisant face à des bactéries renouvelées chaque jour. Il semble que les coûts associés à la virulence et à la résistance conduisent à un équilibre stable entre les phages infectieux et non infectieux ainsi qu’entre les bactéries sensibles et résistantes, et peuvent rapidement limiter le taux adaptatif dans notre système.
Contexte
Les bactériophages, ou phages, et les bactéries sont des micro-organismes ubiquistes ; leur nombre total atteint respectivement 10^31 et 10^30 individus sur Terre. Dans les écosystèmes aquatiques, les phages sont responsables d’environ 10 à 50 % de la mortalité bactérienne. Cette interaction hôte-parasite affecte la stabilité de l’ensemble de l’écosystème. En effet, elle a un impact sur les cycles des ressources avec la libération de nutriments et de débris provenant des cellules lysées, et elle favorise la diversité bactérienne à la fois par des transferts horizontaux de gènes médiés par les phages et par des cycles médiés par les phages dans les densités de population.
En conséquence, la virulence des phages induit une forte pression sélective sur les populations bactériennes, favorisant celles qui deviennent résistantes aux phages. Pour comprendre la dynamique des populations bactériennes, il est nécessaire de prendre en compte l’évolution potentielle de la résistance bactérienne aux phages et la manière dont ces résistances peuvent contrecarrer ces résistances. Bien que les études dans la nature tendent à être plus populaires, depuis près de trente ans, les interactions phages-bactéries ont été étudiées à la fois par l’évolution expérimentale en laboratoire dans des environnements très contrôlés et par des études génétiques. Il a été démontré que certains gènes impliqués dans la résistance aux phages, tels que malT, sont soumis à une sélection diversifiée dans la nature, ce qui suggère une action récurrente des phages sur de tels gènes et, vraisemblablement, des cycles de coévolution des deux partenaires. Le résultat de la coévolution dépend vraiment des organismes étudiés. Par exemple, Escherichia coli B peut supprimer le récepteur T5 sans coût dans des conditions de laboratoire et donc l’extinction des phages suit directement l’émergence de cette résistance. À l’inverse, E. coli B et T4 peuvent coexister sur une longue période. Parmi les systèmes expérimentaux bactéries-phages testés, seulement quelques-uns ont semblé suffisamment stables pour étudier la coévolution, les phages et les bactéries évoluant pour contrecarrer les changements survenant chez l’autre.
La sélection diversifiée observée sur les gènes impliqués dans les interactions hôte-parasite n’est pas limitée aux systèmes bactéries-phages, on la retrouve également lorsqu’il s’agit d’un système immunitaire ou de gènes de résistance des plantes. Dans un cadre encore plus général, toute interaction antagoniste biotique (prédateur-proie ou même conflits sexuels au sein d’une espèce) est censée entraîner une évolution accélérée. Comme l’a illustré Van Vaalen en 1973 avec sa métaphore de la Reine Rouge, la composante biotique de l’environnement est susceptible de conduire la plupart de la sélection pour le changement au sein d’un organisme. Une hypothèse sous-jacente à une telle vision est qu’environnement constant, le taux d’évolution moléculaire devrait être plus lent et moins ciblé que dans un système où des interactions antagonistes biotiques sont présentes.
Pour tester une telle hypothèse, nous avons développé un nouveau système expérimental de coévolution bactérie-phage qui bénéficie des connaissances massives accumulées sur les deux partenaires : E. coli et PhiX174. Ce système est particulièrement pertinent pour mesurer le taux d’évolution car le génome du phage est si petit (5386 pb) qu’il peut être facilement entièrement séquencé après l’évolution. Pour simuler un environnement biotique changeant, nous avons laissé évoluer les bactéries et les phages ensemble (traitement COEV), tandis que pour simuler un environnement biotique constant, nous avons choisi de remplacer chaque jour la population bactérienne des tubes de culture par une population bactérienne ancestrale fraîche, exposant ainsi la population de phages à une évolution directionnelle (DIREV). Nous montrons que la plupart des adaptations se sont produites dans les gènes impliqués dans l’interaction hôte-parasite, mais nous n’avons pas pu détecter de différences claires dans les taux d’évolution entre les deux conditions expérimentales. Nous avons donc cherché la dynamique des facteurs de coévolution qui pourraient expliquer cette observation.
Méthodologie
Souches
PhiX174 (DSMZ4497) et son hôte de référence E. coli C (DSM13127) ont été obtenus de l’institut allemand DSMZ. Une solution stock de PhiX174 a été préparée sur E. coli C, filtrée deux fois et conservée à 4°C (10^9 PFU/mL).
Protocoles d’évolution
Le premier jour, 12 tubes (50 mL) ont été remplis avec 10 mL de bouillon Luria Casein (LC) dilué 4 fois dans MgSO4 (10^-2 M), supplémenté avec du Jus Infectieux (IJ) (10 mL/1 L de la solution stock : CaCl2-0.5 M et MgCl2-1 M)(10^6). Nous avons constaté que la coexistence d’E. coli C – PhiX174 dure plus longtemps dans ce bouillon dilué (données non montrées). Dans chaque tube, E. coli C (10 μL de la culture nocturne à partir d’une colonie unique) a été inoculé avec PhiX174 (10 μL de la solution stock, soit 10^7 PFU, Coefficient d’Infection (COI) : 1). Les tubes ont été incubés à 33°C toute la nuit avec une agitation constante de 130 tr/min ; 6 tubes ont suivi le protocole DIREV (E1 à E6), et 6 le protocole COEV (C1 à C6).
Dans le traitement DIREV, chaque jour, 1 mL de la population a été centrifugé à 10 000 tours par minute (RPM) pendant 5 minutes avec 50 μL de chloroforme, pour se débarrasser des bactéries. Ensuite, 10 μL de ce surnageant ont été inoculés dans un nouveau tube (10 mL 1/4X LC avec IJ) avec 10 μL d’une nouvelle culture nocturne d’E. coli C. Dans le protocole COEV, 10 μL de la population ont été directement inoculés dans un nouveau tube (10 mL 1/4X LC avec IJ). Les 12 tubes, ainsi qu’un tube témoin rempli uniquement de bouillon pour tester les contaminations, ont été incubés à 33°C toute la nuit avec une agitation constante de 130 tr/min.
Stockage
Nous avons collecté chaque jour 500 μL de chaque population et les avons mélangés avec 500 μL de glycérol à 80 % pour le stockage à -20°C.
Coexistence de bactéries et de phages
Chaque jour, la densité optique (OD) des 12 populations a été mesurée à 600 nm. Nous considérions que les bactéries étaient présentes dans la population lorsque l’OD était supérieure à 0.01. Afin de déterminer si les phages étaient toujours présents ou non, nous avons testé le pouvoir de lyse des différentes populations sur l’ancêtre E. coli C. Une couche d’E. coli C a été réalisée sur des boîtes de Petri carrées LC, en versant 9 mL d’agar LC mou (agarose 7.5g/L LC) avec 30 μL de IJ et 300 μL de culture nocturne d’E. coli C(10^6). Nous avons déposé 5 μL des 12 populations différentes sur la couche d’E. coli C. Nous considérions que les phages étaient présents lorsqu’une zone claire apparaissait après une nuit d’incubation à 33°C. Certaines populations ont été titrées plus précisément sur l’hôte ancêtre (1er jour ou D1, D3, D5, D8, D10, D13, D15, D18 et D20).
Isolation des bactéries dans le traitement COEV
Trois populations suivant le protocole COEV (C1, C2 et C3) ont été semées chaque jour sur des boîtes de Petri. Les boîtes ont été incubées à 33°C pendant la nuit et 10 colonies distinctes par population, choisies au hasard, ont été semées à nouveau et inoculées dans LC pour le stockage au glycérol à -20°C.
Test de résistance ou de susceptibilité
Les clones bactériens isolés chaque jour dans C1, C2 et C3 ont été testés pour leur résistance ou susceptibilité aux phages ancêtres et coévolués. Chaque culture nocturne (300 μL) a été mélangée avec 3 mL d’agar mou avec 30 μL de IJ, versée dans une petite boîte de Petri, et nous avons déposé des gouttelettes de 2 μL sur les plaques de la solution stock de PhiX174. Les clones C1 isolés le D1, D3, D5, D8, D10, D12, D14 et D16 ont été mélangés avec 3 mL d’agar mou complété avec 30 μL de IJ, versés sur une boîte de Petri LC et nous avons déposé des gouttelettes de 2 μL de chaque génération de population entière de C1 (en considérant que les bactéries résistantes dans ces populations stockées ne biaiseraient pas les résultats).
Nous avons conclu que les bactéries étaient sensibles lorsque la goutte de phages était associée à une inhibition de la croissance bactérienne après une incubation nocturne à 33°C. Lorsque la zone de la goutte n’était pas différenciable de la couche bactérienne, nous considérions que les bactéries étaient résistantes.
Courbe de croissance bactérienne
Les clones bactériens isolés le D20 de C1, C2 et C3, ont été inoculés dans du LC frais dans une boîte à puits profonds de microplaque pendant la nuit, avec agitation à 37°C. Ensuite, nous avons mis 10 μL de ces cultures avec 190 μL de LC supplémenté avec IJ, dans l’incubateur (TECAN®, infinite 200, Autriche) pour une courbe de croissance de 18 heures à 37°C et agitation régulière.
Concentrations de phages
Pour évaluer l’évolution de la concentration des phages au cours des deux protocoles, nous les avons titrés sur l’hôte ancêtre E. coli C, en utilisant les stocks de solutions glycérolées au jour D1, D3, D5, D8, D10, D13, D15, D18 et D20. Nous avons dilué ces solutions dans du LC frais (dilution de 10^-1 à 10^-6), et déposé des gouttelettes de 2 μL sur une couche d’E. coli C.
Isolation des phages le 20e jour
Le surnageant des 12 populations du D20 a été mélangé avec l’ancêtre E. coli C à différentes dilutions. Cinq plaques de lyse (A à E) ont été choisies au hasard par population, diluées dans un tube Eppendorff de 1 mL LC, vortexées avec 50 μL de chloroforme, et centrifugées pendant 5 minutes, puis le surnageant a été stocké à 4°C. De la même manière, nous avons isolé 5 plaques de lyse de la solution ancêtre de PhiX174 pour un contrôle non évolutif.
Séquençage
Nous avons séquencé quelques phages isolés le D20 : 4 évolués (E2A, E3A, E4A, E6A), 2 coévolués (C1A, C6A) et 1 ancêtre (PhiXA). Nous avons amplifié leur génome avec deux réactions de polymérase en chaîne (PCR), PCR1 avec les amorces 16(+) et 2629(-) et PCR2 avec les amorces 184(-) et 2494(+). Le signe (+) signifie que les amorces ont la même séquence que PhiX174, le signe (-) que les amorces sont le complément inverse de la séquence de PhiX174. Le nombre correspond à la première base de l’amorce dans PhiX174, toutes les amorces ont une longueur de 20 pb. Nous avons choisi 10 amorces pour le séquençage : 674(-), 1106(-), 1629(-), 2181(-), 2629(-) pour PCR1 et 3127(-), 3637(-), 4052(-), 4602(-), 5126(-), 184(-) pour PCR2. Les séquences obtenues ont été analysées avec les logiciels Bioedit et ClustalW.
Taille des plaques de lyse des phages le 20e jour
Les stocks de solutions des 50 phages isolés le D20 (il n’y avait pas de phages dans C2 et C3 le D20), ainsi que ceux des ancêtres PhiX174, ont été titrés sur E. coli C, comme décrit ci-dessus. Nous avons étalé quelques dilutions de ces solutions dans un mélange d’agar en surface (environ 10 à 50 plaques par boîte de Petri). Nous avons réalisé ce protocole en deux réplicats. Nous avons mélangé les boîtes de Petri dans l’incubateur, à 33°C, pour ne pas tenir compte de la position des boîtes et de l’influence du radiateur. Le lendemain, nous avons pris des photos des boîtes avec Biocapt, et les avons analysées avec un logiciel fait maison qui nous a donné la taille (en pixels) et le nombre total de plaques par boîte de Petri.
Courbes de lyse des virus séquencés
Les sept virus séquencés ont été inoculés avec E. coli C, en 6 réplicats, dans une microplaque, avec un COI de 10^-5, dans du LC supplémenté avec IJ. Il a été incubé à 37°C dans le TECAN, la densité optique à 600 nm a été mesurée régulièrement, et la plaque a été agitée.
Coexistence de bactéries et de phages
Nous avons d’abord souhaité tester la possibilité de coexistence de phiX174 et d’E. coli C, étant donné que de nombreux systèmes bactéries-phages précédents ont échoué à cette étape. Comme la disponibilité des ressources pourrait être un paramètre important, nous avons d’abord réalisé plusieurs séries d’expériences avec diverses concentrations de ressources, et nous avons constaté que le bouillon Luria Casein (LC) dilué 4 fois dans un milieu salin fournissait le taux le plus élevé de coexistence des deux partenaires (données non montrées). Les concentrations bactériennes de chaque population ont été mesurées chaque jour, ainsi que la présence des phages. Pendant le traitement DIREV, nous avons observé que les phages étaient maintenus jusqu’au D20, avec quelques extinctions ponctuelles des bactéries (Figure IA). Dans le traitement COEV, dans 4 des 6 populations, les bactéries et les phages coexistaient toujours au D20 (C1, C4, C5 et C6), tandis que les phages s’éteignaient vers le D11 ou D12 dans les populations C2 et C3.
Pour vérifier si des changements génétiques liés à la coévolution antagoniste s’étaient produits chez les deux partenaires, nous avons d’abord réalisé un test simple. Nous avons vérifié dans la population C1 si les bactéries évoluées étaient toutes résistantes au phage ancestral. Comme cela s’est avéré être le cas, cela suggère que le phage et les bactéries ont coévolué dans une certaine mesure. Taux d’évolution du virus coévoluant continuellement avec les bactéries ou non Pour évaluer si la présence de la coévolution avait accéléré le taux d’adaptation moléculaire, nous avons séquencé le génome évolué et le génome ancestral. La séquence du virus ancêtre diffère de la séquence de Sanger avec 7 mutations (données non montrées). En raison de la petite taille des plaques et de l’extinction de deux populations de phages, nous n’avons pu séquencer que 2 lignées COEV et 4 lignées DIREV (nous avons voulu éviter de plaquer le phage à haute densité pour éviter l’évolution de novo sur la boîte). Nous avons d’abord identifié les mutations qui étaient apparues au cours de l’expérience, et étudié l’impact de ces mutations sur la composition en acides aminés des protéines virales (Tableau I). Nous avons trouvé 25 substitutions différentes, pour un total de 41 événements de mutation. La plupart des substitutions (80%) sont apparues uniquement dans l’un ou l’autre traitement : 11 dans les virus évolués DIREV et 9 dans les virus COEV. Certaines (5, 20%) sont apparues dans les deux traitements. Certaines substitutions (7, 28%) ont émergé en parallèle dans différentes lignées. Outre les mutations parallèles que nous avons observées, certaines substitutions (6, 24%), émergeant dans des isolats différents ou non, avaient déjà été décrites dans la littérature. Cette information est importante car elle implique que des substitutions uniques pourraient être sélectionnées dans des lignées évoluées indépendantes. T1354A et G2015C étaient apparues dans un protocole de coévolution en chemostat avec alimentation continue en E. coli C ; G1319A avait été considérée comme une mutation compensatoire. Enfin, C1460G et C2085T avaient été observées dans un protocole d’adaptation à des hôtes alternés (E. coli C et Salmonella). Dans les deux traitements, comme prévu selon un modèle de coévolution, les mutations étaient fortement regroupées, touchant uniquement les gènes impliqués dans la capside ou le procapside. En effet, bien que le gène F représente 24% du génome, il renfermait plus de 70% de toutes les mutations (test exact de Fisher p=5 10-12). Au total, les gènes de la capside, qui représentent 56% du génome, portaient 84% des mutations (test exact de Fisher p=5 10-5). Comme attendu pour une évolution moléculaire entraînée par la sélection, la plupart des mutations étaient non synonymes, avec un rapport total de mutations synonymes sur non synonymes de 1,75 et 1,5 pour COEV et DIREV respectivement. Cependant, quatre des cinq mutations survenant dans le gène D étaient synonymes. Cela est cohérent avec l’observation faite par Burch et Fane selon laquelle ce gène est le plus conservé dans la famille Microviridae de PhiX174, mais n’explique pas pourquoi 5 mutations ont été trouvées dans ce gène par rapport à 0 dans le gène A, qui est trois fois plus grand (test exact de Fisher p=6 10-4), ce qui suggère que D pourrait être un locus avec une forte pression sélective sur les structures secondaires de l’ADN. Bien que cette répartition des mutations à travers le génome renforce l’idée de la coévolution, il n’y avait pas de puissance pour détecter des différences dans les motifs ou les taux entre les deux traitements. Même si les lignées DIREV avaient un nombre légèrement inférieur de mutations par rapport à COEV, la différence n’était pas significative. Ainsi, de manière surprenante, nous pouvons conclure que même si les phages du traitement COEV coévoluaient continuellement avec les bactéries, leur taux d’évolution n’est pas significativement augmenté par rapport aux phages du traitement DIREV, comme nous l’avions prévu. Nous avons cherché des facteurs pouvant contribuer à cette observation. Le taux d’évolution adaptative des deux partenaires est directement corrélé au nombre de mutations bénéfiques générées à chaque génération et à leur effet sur la fitness. Nous avons donc étudié les tailles de population et l’intensité de la sélection imposée à chaque antagoniste.
Taille de la population virale
Nous nous sommes d’abord concentrés sur les tailles de population virale. Même si nous ne prenons pas en compte les populations C2 et C3 où les phages s’éteignent vers le D11 ou D12, il est apparu que la concentration virale était toujours significativement plus faible (test t significatif, p<0,05) dans le traitement COEV après le jour 8 par rapport au traitement DIREV. Cette différence pourrait être un effet confusionnel dans le taux d’évolution, mais il est difficile de quantifier son importance sans un modèle intégré approprié de coévolution, car cela affectera à la fois la capacité de la population de phages à répondre à la sélection et diminuera également la pression sélective imposée par les phages sur les bactéries.
Coût de la résistance/virulence
Pour que les interactions biotiques entraînent une adaptation plus rapide, il faut que la pression sélective associée aux interactions biotiques soit plus forte que l’avantage sélectif des mutations bénéfiques dans un environnement constant. Lorsqu’on parle de résistance et de virulence, on envisage des pressions sélectives très intenses. Cependant, si la pression sélective imposée par chaque partenaire sur son antagoniste est réduite, le taux de coévolution peut être réduit. Une telle réduction du taux de coévolution peut être déclenchée par un gain limité associé aux nouvelles mutations bénéfiques et/ou par une pression sélective atténuée imposée à l’antagoniste. Par exemple, comme suggéré dans la plupart des modèles de gène pour gène de l’évolution, si une résistance étendue ou une plage d’hôtes élargie est associée à un coût, chaque allèle correspondant se propagera plus lentement dans les populations que le précédent. Le système peut éventuellement atteindre un équilibre polymorphe lorsque le coût de l’allèle de résistance/virulence ultérieur est de même ordre que son avantage associé. Pour tester si un tel processus existait dans notre système expérimental, nous avons utilisé deux approches. D’une part, nous nous sommes concentrés sur la virulence sur le génotype bactérien ancêtre des phages isolés au D20. D’autre part, nous avons suivi l’évolution de la résistance et de la plage d’hôtes des phages à travers la population COEV C1 pour voir si les interactions entre les phages et les bactéries étaient compatibles avec l’émergence d’une pression de sélection réduite induite par les phages sur la population bactérienne.
Virulence et productivité des bactériophages coévolués sur l’hôte ancêtre
L’impact des virus coévolués au D20 sur la courbe de croissance de l’hôte ancêtre a été mesuré et comparé à l’impact du bactériophage ancêtre et des virus D20 du traitement COEV. Les bactéries ancestrales inoculées sans phages croissent de manière exponentielle. Dans les puits où les phages ont été ajoutés, la concentration bactérienne a d’abord augmenté, puis a chuté de manière spectaculaire. Selon la forme des courbes de lyse, le virus ancêtre (la ligne verte) était le virus le plus virulent sur la souche de référence. En effet, nous avons observé une virulence significativement réduite pour la plupart des virus D20, par rapport à l’ancêtre, mais curieusement, nous ne pouvons pas clairement distinguer les virus du traitement DIREV (lignes noires) de ceux du COEV (lignes rouges). Cependant, cette observation était cohérente avec l’existence d’un coût associé aux mutations sélectionnées dans notre traitement. Pour évaluer davantage ce coût, nous avons plaqué sur l’hôte ancêtre des populations issues de 5 clones isolés de chacune des populations virales au D20. En effet, la croissance des plaques peut être un test plus sensible que le test précédent, car elle reflète le produit de nombreuses infections supplémentaires. Encore une fois, les populations virales DIREV et COEV avaient une productivité significativement diminuée, mesurée par la taille des plaques, par rapport au bactériophage ancêtre (les deux ANOVA sont significatifs, p<10-15). Cette fois, cependant, il y avait une différence significative entre les traitements (ANOVA significatif, p<10-15). Les virus DIREV étaient plus adaptés que les virus COEV qui avaient une taille de plaque considérablement réduite sur l’hôte ancêtre. Cela suggère que la perte de productivité est plus élevée lorsque les virus sont confrontés à des bactéries coévoluant continuellement, comme si les phages modifiaient de manière plus drastique les paramètres optimaux de l’infection sur la souche sauvage d’E. coli C. Ces deux résultats étayent l’hypothèse d’une virulence réduite des bactériophages coévolués (au moins, sur l’hôte ancêtre).
Émergence de la résistance dans les populations bactéries-phages en coévolution continue
Pour comprendre dans quelles conditions cette réduction potentielle de la virulence du phage était observée, nous avons décidé d’examiner plus attentivement la population C1 pour comprendre comment les phages et les bactéries pouvaient s’infecter mutuellement ou résister l’un à l’autre. Nous avons utilisé des populations de phages des 20 premiers jours et les avons confrontées à 10 clones indépendants échantillonnés à partir des populations bactériennes des jours D1, D3, D5, D8, D10, D12, D14 et D16. Lorsque la présence de phages était associée à une inhibition de la croissance bactérienne, on disait que les bactéries étaient sensibles à la génération de phages. Sur les 1600 confrontations de ce type, nous avons identifié 30 motifs différents de résistance bactérienne (y compris l’ancêtre) et 18 motifs de plage d’hôtes pour les populations de phages. Après avoir trié les rangs et les colonnes de la matrice, nous avons identifié une matrice d’infection globalement, mais pas exclusivement compatible avec un modèle de gène pour gène, bien que nous ne sachions pas combien de mutations sont nécessaires pour passer d’un motif à un autre. En effet, la diagonale inférieure de la matrice est pleine de croisements infectieux, ce qui signifie qu’il tend à y avoir une hiérarchie à la fois dans la résistance et dans la plage d’hôtes : principalement, le phage à large plage d’hôtes peut infecter certaines des bactéries les plus résistantes. Dans un tel système, s’il n’y a pas de coût associé à une plage d’hôtes plus large ou à la résistance, on s’attendrait à une sélection de la plage d’hôtes la plus large pour le phage et de la résistance la plus élevée pour les bactéries, dans une course aux armements continue. Lorsque nous avons suivi l’évolution de la plage d’hôtes globale et de la résistance, nous avons cependant été surpris de voir les bactéries les plus résistantes et les phages à plage d’hôtes la plus large émerger tôt dans la coévolution, puis voir une résistance et une plage d’hôtes intermédiaires prendre le relais. Cela est cependant compatible avec l’existence de coûts pour la résistance et une plage d’hôtes plus grande. En raison d’un taux de mutation variable, les bactéries les plus résistantes et à plage d’hôtes plus grande ont peut-être émergé en premier, mais ensuite, en raison de leur coût, des phénotypes intermédiaires avec un coût moindre ont pu se propager. L’image est donc plus complexe que prévu par des modèles simples, mais le maintien d’une grande fraction de bactéries sensibles suggère que la charge imposée par la prédation des phages est beaucoup réduite plus tard dans l’évolution. De plus, la forte variabilité du phénotype de résistance présente dans la population suggère que la population bactérienne ne subit pas une sélection complète dans laquelle un nouvel allèle de résistance envahirait toute la population, mais plutôt un équilibre entre ces allèles de résistance probablement entraîné par leur coût. Ainsi que suggéré, nous pensons que la pression sélective induite par la prédation des phages n’est peut-être pas aussi forte tout au long de toute la coévolution et peut donc modérer l’accélération potentielle de l’adaptation suggérée.
Homogénéité de la population bactérienne dans les populations du traitement de contrôle
Enfin, il est à noter que ce que nous avons utilisé comme contrôle d’un environnement constant pourrait ne pas être le contrôle idéal. Dans le traitement DIREV, la plupart des jours, certains clones bactériens résistants sont isolés des populations juste avant le transfert des virus dans un nouveau milieu de culture. Cela signifie que même dans ce cadre, des génotypes résistants ont émergé et ont remplacé les génotypes sensibles naïfs. De plus, étant donné que chaque jour nous inoculions des bactéries fraîches, différentes mutations de résistance auraient pu être sélectionnées, contredisant notre hypothèse selon laquelle la population bactérienne était génétiquement homogène dans le traitement de contrôle. Nous aurions pu choisir une durée de cycle plus courte pour éviter une telle émergence de résistance. Cependant, le temps de cycle doit être suffisamment long pour permettre 6 à 10 générations bactériennes, ce qui peut nécessiter au moins 4 à 5 heures avec E. coli C qui n’est pas une souche d’E. coli à croissance rapide. Cependant, des cycles de 4 à 5 heures ne sont pas entièrement compatibles avec le travail de laboratoire, le stockage des populations entre les cycles devient donc un problème car il peut avoir un impact sur la dynamique de l’interaction bactérie-phage. Remarques finales Dans l’ensemble, nous avons observé une évolution rapide dans les deux conditions expérimentales qui était concentrée dans une partie très spécifique du génome : la protéine F qui est connue pour médier la reconnaissance de l’hôte. Nous n’avons pas pu observer un taux d’évolution plus rapide dans le traitement COEV, mais cela peut être dû au fait que le contrôle n’était pas parfait et au fait que les tailles de population virale étaient plus faibles en coévolution qu’elles ne l’étaient dans les populations de contrôle. Enfin, nous suggérons à travers notre système que la résistance et la plage d’hôtes étendue peuvent avoir un coût qui peut finalement conduire à une pression sélective réduite imposée par le phage sur la population bactérienne, ce qui peut à son tour entraîner une évolution moins dynamique. Le présent travail présente un nouveau système expérimental de coévolution bactérie-phage qui semble assez riche car la plupart des populations de virus avaient des motifs différents de plage d’hôtes et jusqu’à 30 phénotypes de résistance différents ont été isolés parmi les bactéries. Comme les deux partenaires sont particulièrement bien caractérisés et peuvent être génétiquement modifiés, ce système peut être puissant pour élucider les bases moléculaires d’une interaction bactérie-phage.
Nicolas Dumont
Annonces PARTENAIRES
Centre de formation CFA - Certifié Qualiopi Diplômes RNCP et certifications RS.
3axes academy : Formation, Innovation, Excellence
VOIR NOS FORMATIONSOffrez-vous cette emplacement publicitaire